Un nouvel outil moléculaire permet de modifier les génomes à volonté, chez tous les êtres vivants.
Christelle Gally consacre sa vie professionnelle à un ver. Un petit nématode transparent d’environ un millimètre, organisme modèle des biologistes : Caenorhabditis elegans. La chercheuse de l’Institut de génétique et de biologie moléculaire et cellulaire de Strasbourg en scrute les gènes, crée des mutants, observe les effets de ces manipulations, afin de mieux comprendre ce que l’on nomme la reprogrammation cellulaire : la transformation d’une cellule de la peau en neurone, par exemple. Désir de percer un mécanisme fondamental du vivant. Mais aussi espoir d’y trouver une application thérapeutique chez l’homme. « Faire remarcher les paralysés », avoue-t-elle.
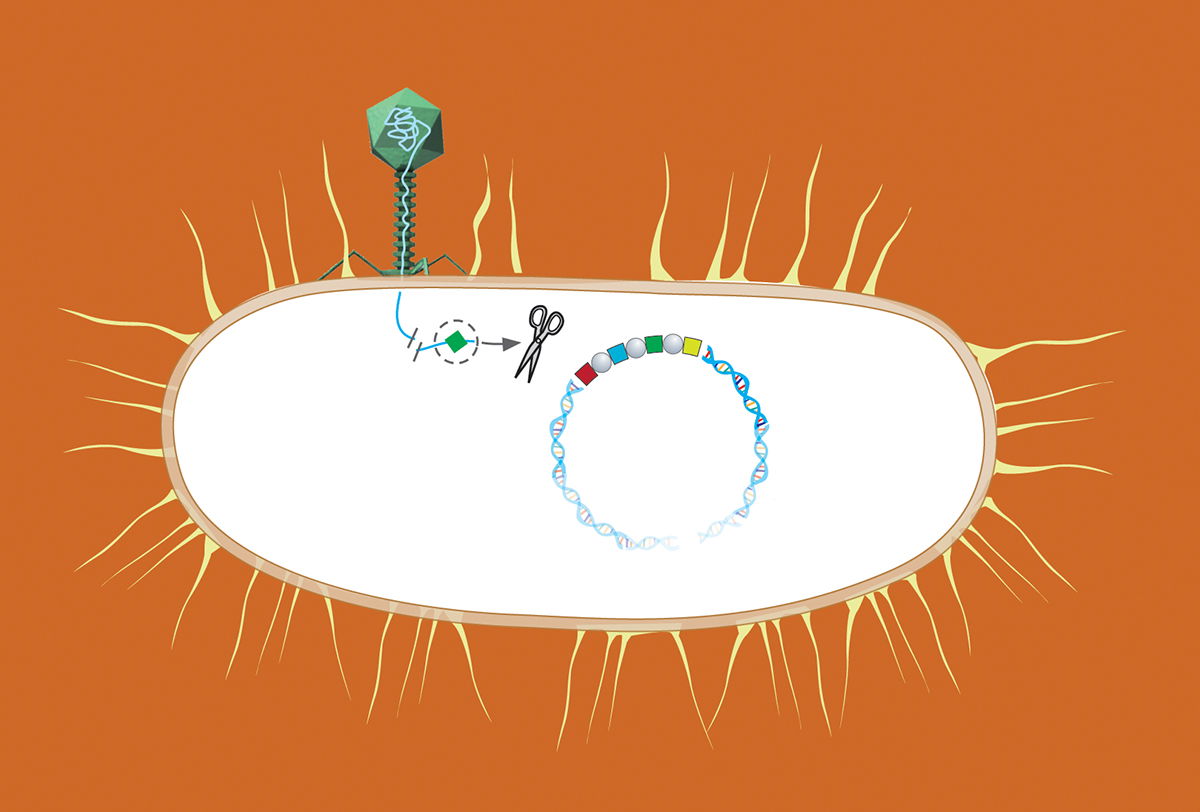
Dans cette quête, Christelle Gally a longtemps fait avec les moyens du bord. « On créait des mutants de façon aléatoire, on regardait ceux qui étaient paralysés et on cherchait quel gène avait changé. Ou alors on choisissait un gène que l’on modifiait et on en observait les effets. On avait inventé une méthode, on en était fiers. Et puis Crispr-Cas9 est arrivé. Ce qui prenait des mois prend désormais une semaine, ce qui était approximatif est devenu fiable, ce qui était temporaire est devenu stable, ce qui était cher est bon marché. » Elle poursuit : « Quand je m’y suis mise il y a deux ans, j’étais une pionnière dans la communauté C. elegans. Aujourd’hui, je ne connais pas un labo qui ne fasse pas du Crispr. Pour nous, c’est une révolution. »
Le « nous », dans sa bouche, désigne sa « communauté ». Pourtant, c’est une révolution bien plus vaste qui se cache derrière ce mystérieux acronyme, Crispr-Cas9. Au-delà des adeptes de C. elegans, et d’autres animaux modèles, tous les biologistes se sont jetés sur l’invention. Mais aussi des médecins, et depuis peu, des philosophes et des juristes… Du secret des laboratoires, le « couteau suisse de la génétique » a gagné la tribune des académies scientifiques du monde entier, des réunions internationales ad hoc, des parlements nationaux…
C’est que Crispr-Cas9 appuie là où ça fait mal. Qu’est-ce qu’un organisme génétiquement modifié ? Par sa simplicité et sa nature, le nouvel outil moléculaire a d’ores et déjà rouvert le débat. Peut-on modifier ou éliminer l’ensemble d’une espèce (par exemple le moustique responsable du paludisme) pour sauver des centaines de milliers de vies humaines ? Par sa précision et son efficacité, il impose la réflexion. Mieux, ou pire : pour la première fois de son histoire, l’humanité va pouvoir modifier sa propre nature, réécrire l’ADN de notre espèce. Alors, tous grands, blonds aux yeux bleus ?
« Un outil d’édition du génome, presque universel, avec de telles implications, jamais je n’aurais pu l’imaginer »
Francis Mojica
chercheur au laboratoire de l’université d’Alicante (Espagne)
Dans son laboratoire de l’université d’Alicante, Francis Mojica ne cache pas son vertige. « Je pensais qu’il y aurait des applications à mon travail, en agriculture, peut-être en médecine, affirme le biologiste. Mais qu’il aboutirait à un outil d’édition du génome, presque universel, avec de telles implications, jamais je n’aurais pu l’imaginer. » Aurait-il affirmé l’inverse qu’on ne l’aurait pas cru. Car s’il est désormais de coutume d’attribuer à Mojica la pose des premières pièces du puzzle, c’est d’un morceau de ciel dans un vaste paysage qu’il s’agit alors. Ou plus exactement d’un coin de mer.

Nous sommes en 1989. Tout juste sorti du service militaire, l’étudiant en thèse âgé de 26 ans s’attache à comprendre ce qui rend Haloferax mediterranei, un organisme unicellulaire de la famille des archées, si résistant aux hautes concentrations salines des marais voisins. Le séquençage du génome en est alors à ses balbutiements, mais l’équipe d’Alicante se lance dans l’entreprise. Mojica découvre une particularité qui éveille sa curiosité : certes, il y a là des séquences d’ADN assez longues, correspondant à des gènes, comme dans tous les organismes ; mais aussi d’autres, beaucoup plus courtes, qui – là tient la nouveauté – se répètent plusieurs fois. A quoi correspondent ces répétitions ? « Nous n’en avions aucune idée, admet Mojica. Mais, en plongeant dans la littérature, je me suis aperçu que des Japonais avaient déjà fait la même observation sur la bactérie modèle E. coli. Une même structure chez deux organismes très éloignés : il y avait des chances que ce soit plus largement présent et que ça ne soit pas anodin. »
L’ADN des bactériophages
Et voilà Francis Mojica lancé dans l’œuvre de sa vie.Comme nombre de jeunes « docteurs », il se rend à Oxford pour une « parenthèse » de deux ans consacrée à l’étude d’une protéine d’E. coli. « Mais je ne supportais pas la pluie. » Retour à Alicante, donc. Et à Crispr. Qui ne s’appelle d’ailleurs pas Crispr. Lui a dénommé sa trouvaille SRSR (Short Regularly Space Repeat). Aux Pays-Bas, un autre jeune chercheur, Ruud Janssen, a fait des observations similaires. Il a baptisé la structure « Spider ». Les deux universitaires échangent des mails. Finalement, Mojica propose Crispr, pour « Clustered Regularly Interspaced Short Palindromic Repeats », ce qui pourrait se traduire par « groupement d’éléments palindromiques courts répétés et régulièrement espacés ». « Ça sonnait bien, c’était complet, nous nous sommes mis d’accord », se souvient Mojica – ignorant alors que l’acronyme sonne encore mieux en anglais où il signifie « plus croquant », « précis », « net ».
Janssen publiera le nom en 2002. En passant en revue les génomes connus, l’Espagnol et le Hollandais découvrent des Crispr chez pas moins de quarante espèces de bactéries et d’archées. Une approche bio-informatique que Francis Mojica n’a pas vraiment choisie. « En rentrant d’Oxford, mon labo avait déménagé. Il n’y avait ni argent ni vraiment de place pour moi. Juste un ordinateur. Alors, j’ai étudié le phénomène sur ce versant-là. » En 2002, avec le premier financement obtenu, il analyse les séquences répétées dans les Crispr de différentes souches de bactérie E. coli. Puis il s’attaque aux groupes de bases qui les séparent. Et c’est en comparant ces « espaceurs » à l’ADN des bactériophages – ces virus qui attaquent les bactéries – qu’il fait « sa » découverte. « Je n’oublierai jamais. C’était en août 2003, il faisait horriblement chaud, je passais mon temps au labo, car c’était calme et climatisé. Dans une souche, j’ai vu un premier espaceur qui correspondait à un phage contre lequel la souche était justement résistante. Tout de suite, j’ai pensé que ça pouvait être un système immunitaire adaptatif. J’ai foncé à la plage dire à ma femme que j’avais découvert un truc incroyable. Je n’avais pas la preuve, mais j’en étais sûr. Je lui ai même parlé du Nobel. »
Immunité adaptative
Incroyable, en effet. Car si les mammifères disposent d’une telle immunité adaptative (les fameux anticorps), les organismes unicellulaires en sont, pense-t-on alors, dépourvus. Le scientifique espagnol passe en revue les Crispr disponibles et retrouve, à cinquante reprises, cette même « correspondance » entre un espaceur et une portion d’ADN phagique. Seul problème : ce que l’ADN suggère, il ne parvient pas à le reproduire en mettant en présence la bactérie et son phage. En novembre 2003, le journal Nature rejette son article. En janvier, Proceedings of the National Academy of Sciences (PNAS) le repousse à son tour. Deux autres revues, à chaque fois un peu moins prestigieuses, déclinent. « Un des reviewers m’a avoué plus tard qu’il ne pouvait simplement pas y croire »… Finalement, c’est dans une publication mineure, Journal of Molecular Evolution, et après moult corrections, que l’article est publié, le 1er février 2005.

Il était temps ! Car deux autres équipes françaises planchent sur le sujet. A l’université d’Orsay, Gilles Vergnaud et Christine Pourcel poursuivent de tout autres motivations. Vergnaud s’est vu passer commande par le ministère de la défense du classement de bactéries de la peste, Yersinia pestis. « C’était après le 11 Septembre et les envois d’enveloppes d’anthrax, se souvient le chercheur. Il fallait se préparer à une attaque biologique. On nous avait confié l’ADN de soixante et une souches, issues d’une épidémie au Vietnam entre 1964 et 1966. » La comparaison est rapide, les empreintes génétiques sont toutes identiques sauf sur une zone : les Crispr. Les deux scientifiques décident donc d’analyser en détail cette petite zone sur leurs soixante et un échantillons, toujours dans l’optique d’un classement.
« Chasse gardée »
Mais c’est autre chose qu’ils découvrent : à savoir qu’avec le temps les séquences Crispr s’enrichissent de nouveaux fragments d’ADN successifs et que ceux-ci correspondent à des bactériophages répertoriés comme attaquant les bacilles de la peste. « Sous nos yeux, nous pouvions voir la construction de l’immunité des bactéries, même si l’expérience avait eu lieu quarante ans plus tôt », insiste Christine Pourcel.
Leur enthousiasme sera douché. Eux n’ont pas visé Nature. « C’est la chasse gardée de quelques équipes bien connues », soupire Christine Pourcel. PNAS ne se montre pas plus accueillant. Trois autres revues rejetteront le texte. Avant que Microbiology n’accepte de le publier, le 1er mars 2005.
C’est cette même revue qui, en septembre, publie le troisième article de la désormais fameuse trilogie de 2005. Il est signé par Alexander Bolotin et Dusko Ehrlich, deux chercheurs de l’Institut national de recherche agronomique (INRA). Leur objet d’étude : Streptococcus thermophilus, la bactérie du yaourt. Les premiers, ils en ont séquencé le génome. Puis l’ont étudié. Et, à leur tour, ils découvrent que les espaceurs des Crispr correspondent à des séquences de bactériophages. « On a alors étudié les génomes de bactéries déjà séquencés et on a retrouvé le même résultat, raconte Dusko Ehrlich. On s’est même rendu compte que plus il y avait d’espaceurs, plus la résistance aux phages augmentait… Nous avons donc écrit qu’il s’agissait probablement d’un système de protection. »
La bactérie « E. coli »
Pour aller plus loin, il faut non plus seulement analyser l’ADN, mais mettre véritablement en présence les bactéries et leurs virus. Tous y songent. Mojica a essayé, en vain. Il comprendra plus tard que, chez la bactérie E. coli, un autre système immunitaire domine Crispr. Vergnaud et Pourcel, de leur côté, demandent au ministère de la défense la possibilité de réaliser les expériences. « Mais on ne manipule pas la peste comme ça, dit Gilles Vergnaud. Il faut des installations spéciales. Le ministère a refusé. » Leurs dossiers devant l’Agence nationale de la recherche connaîtront le même sort. Quant à l’équipe de l’INRA, elle dispose bien de quelques souches de S. thermophilus mais trop peu, et encore moins des collections de phages nécessaires pour conduire l’expérience. Elle fait alors appel aux industriels. Nestlé exige la propriété intellectuelle en cas de découverte ; son concurrent, le groupe de biotechnologie Danisco, refuse. « Nous avons donc renoncé. L’année suivante, nous avons compris », déclare Dusko Ehrlich.
En février 2007, une équipe franco-canadienne conduite par des chercheurs de Danisco publie dans Science un article, qui, cette fois, fait grand bruit : la démonstration expérimentale de l’existence d’un système immunitaire adaptatif chez les bactéries. Crispr-Cas9 sort de la préhistoire. Une nouvelle aventure commence.