Des ciseaux dans le yaourt

Un gros bourg agricole de 3 500 habitants, comme la France en compte tant. Sur l’atlas mondial de Crispr-Cas9, ce système révolutionnaire d’édition des génomes, Dangé-Saint-Romain (Vienne) tient une tout autre place. Une position éminente, même, entre San Francisco, Boston, Vienne et Paris. C’est en effet d’ici qu’entre 2005 et 2007 a été coordonné le programme de recherche qui a apporté la preuve expérimentale de l’existence d’un système immunitaire adaptatif chez les bactFMoiéries, prélude à la révolution Crispr-Cas9, cet outil qui bouleverse aujourd’hui la biologie mondiale.
Pourquoi ici ? « Parce qu’il y avait une laiterie, sourit Philippe Horvath, chercheur au centre de recherche de la multinationale de l’agroalimentaire et de la chimie DuPont. En 1964, un ingénieur y a créé une petite entreprise de ferments qui a grossi, a été rachetée par Rhône-Poulenc, puis par Danisco, avant qu’en 2011 DuPont rachète Danisco. Aujourd’hui, nous exportons partout. Un yaourt sur deux et un fromage sur trois produits dans le monde contiennent des ferments qui viennent de chez nous. »

Le rapport avec le système immunitaire des bactéries ? Pédagogue, Philippe Horvath revient aux bases : « Les ferments, ce sont des bactéries. Sans elles, pas de fromage, le lait reste du lait. Or, les bactéries ont un ennemi mortel : les virus, plus précisément les bactériophages. Ça fait douze mille ans qu’on utilise l’acidification pour conserver les produits laitiers. La technique s’est améliorée : chaque ferment contient plusieurs bactéries et produit sa propre texture, son acidité, son goût… Mais tout ça reste une course de vitesse entre bactéries et phages, à qui s’adaptera plus vite à l’autre. Mon rôle, c’est d’aider les bactéries. »
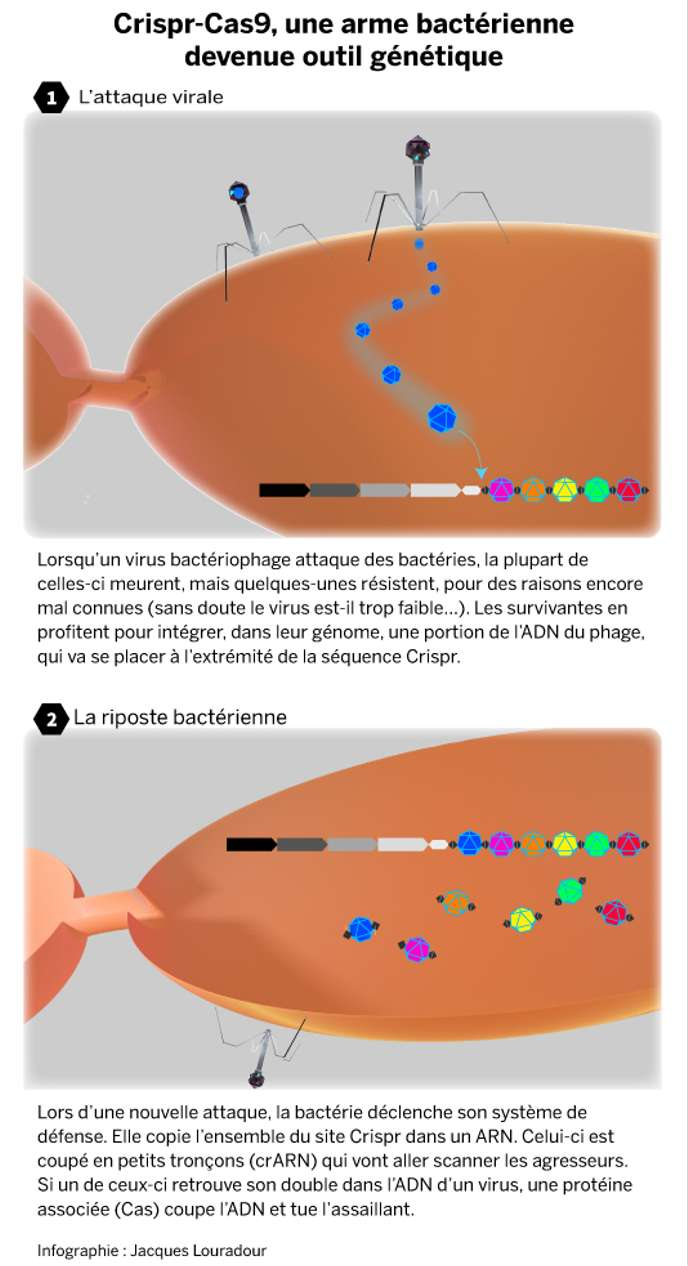
En 2000, le jeune docteur de l’université de Strasbourg débarque ainsi à Dangé-Saint-Romain avec pour mission d’utiliser les outils de la biologie moléculaire pour classer les quelque 13 000 souches bactériennes maison. La tâche est vaste, il tâtonne. Deux ans plus tard, il présente un « poster » lors d’un congrès sur les bactéries lactiques aux Pays-Bas. A côté de lui, un ingénieur de recherche de l’Institut national de recherche agronomique (INRA), Alexander Bolotin, affiche lui aussi son travail : le premier séquençage du génome de la bactérie du yaourt Streptococcus thermophilus. En 2005, il signera un des trois articles suggérant l’existence d’un système immunitaire adaptatif chez les bactéries. « Ils avaient déjà mis en évidence dans l’ADN les séquences répétées du Crispr et, entre elles, les espaceurs, tous différents », se souvient Philippe Horvath.
Le brouillard se lève
Après le rachat par le géant danois des biotechnologies Danisco, le centre de recherche hérite, à l’hiver 2004, d’un séquenceur d’ADN. Philippe Horvath commence donc à établir la carte d’identité génétique des souches bactériennes et des phages conservés au laboratoire. Il sait quelle bactérie résiste à quel phage. Aussi l’évidence s’impose-t-elle : les bactéries résistantes portent dans leur Crispr des séquences correspondant précisément aux phages auxquels elles échappent. Publier immédiatement ? En Espagne et en France, le même constat a déjà été réalisé « in silico », autrement dit à partir de l’ADN conservé dans les bases de données informatiques. « Surtout, nous sommes des industriels, rappelle Philippe Horvath. La priorité, c’était le brevet. » En août 2005, Danisco dépose donc devant l’Office américain des brevets (US Patent and Trademark Office, ou USPTO) une demande d’homologation d’un système d’« utilisation » de Crispr pour offrir aux bactéries une résistance aux phages.

La procédure donne un an à l’entreprise pour étoffer et nourrir d’exemples son invention. La tâche est lourde. Philippe Horvath dispose de l’équipe de Dangé mais aussi d’un jeune scientifique français tout juste embauché par Danisco dans son centre de recherche américain, à Madison (Wisconsin) : Rodolphe Barrangou. Aujourd’hui professeur à l’université de Caroline du Nord, il se souvient : « On a altéré le contenu Crispr de certaines souches bactériennes. Quand on ajoutait l’ADN phagique, on vaccinait la cellule. A l’inverse, quand on le retirait, la bactérie perdait sa protection. Et quand on échangeait les espaceurs entre deux bactéries, on échangeait aussi leur résistance. »
Autant de manipulations qu’il convient de réaliser sur plusieurs souches. L’équipe française comprend qu’elle ne tiendra pas les délais imposés par l’administration américaine. Elle fait donc appel à Sylvain Moineau, professeur de microbiologie à l’université Laval de Québec spécialiste des phages et collaborateur régulier de Danisco. Le brevet définitif est déposé en août. Puis ils s’attellent ensemble à la rédaction d’un article scientifique, transmis en octobre 2006, à la revue Science. « Je n’y croyais pas vraiment, se souvient Rodolphe Barrangou. Un papier sur la bactérie du yaourt, écrit par une boîte privée… Ce que je ne savais pas, c’est que Jill Banfield, une formidable scientifique de Berkeley, unanimement reconnue, avait envoyé un papier sur le même sujet deux semaines avant. Le sien était moins complet. Grâce à elle, le nôtre a été pris… » Et encore… Deux des trois scientifiques indépendants chargés d’évaluer le texte s’opposent à sa publication. L’éditeur offre une seconde chance aux auteurs, qui étoffent leur copie. Un des reviewers bascule. L’article paraît le 23 mars 2007.

Cité, depuis, dans plus de mille publications scientifiques, il marque la « première révolution Crispr », comme le milieu aime à la nommer. Pour les biologistes, c’est la démonstration, deux ans après la formulation de l’hypothèse, de l’existence d’une immunité adaptative des organismes unicellulaires. Pour les généticiens et biochimistes, le coup d’envoi d’une nouvelle épopée : comprendre Crispr. Car si l’acquisition de portions de génome d’un virus a déjà été largement décrite chez nombre d’organismes vivants – de la bactérie à l’homme –, le fonctionnement du nouveau vaccin génétique reste un mystère. Comment la bactérie reconnaît-elle son ennemi ? Comment le combat-elle ?
A travers le monde, une nouvelle communauté voit le jour. Avec ses têtes d’affiche et sa messe annuelle : la réunion Crispr. En 2008, ils sont une trentaine à répondre à l’invitation de Jill Banfield et Rodolphe Barrangou à Berkeley. « C’était le Noël des chercheurs, se souvient Sylvain Moineau. On était comme des enfants devant l’arbre. On découvrait les cadeaux apportés par les autres. » « Etre invité en Californie pour parler de mon petit sujet qui intéressait des gens aussi éminents, c’était incroyable », renchérit l’Espagnol Francis Mojica, auteur, en 2005, du premier article envisageant une immunité acquise des bactéries.
Peu à peu, le brouillard se lève. En 2008, le Néerlandais John van der Oost fait un premier pas. Il montre comment l’ARN messager (qui transporte l’information contenue dans l’ADN) de la bactérie est coupé en petits morceaux (crRNA), qui s’associent à des protéines pour aller chasser les virus ennemis. Il parvient également à programmer une bactérie afin de lui apporter la résistance à un phage donné.
Quelques mois plus tard, deux chercheurs de Chicago, l’Argentin Luciano Marraffini et son directeur de laboratoire, l’Américain Erik Sontheimer, montrent que Crispr répond aux virus mais aussi aux plasmides, ces bouts d’ADN porteurs de traits particuliers, comme la résistance aux antibiotiques. Ils apportent également une preuve indirecte que c’est l’ADN des virus qui est visé par le système, et non l’ARN, comme la plupart des chercheurs le pensaient jusque-là. Dans la conclusion de l’article qu’ils publient dans Science, ils affirment enfin, prophétiques, que Crispr « pourrait avoir une utilité considérable, surtout si le système devait fonctionner hors de son contexte bactérien ».
Le sprint final
Le Canadien Sylvain Moineau a poursuivi sa quête. Dans un premier article, en 2008, il établit l’importance de minuscules morceaux d’ADN qu’il baptise PAM (protospacer adjacent motif). La bactérie en est démunie. Le virus, au contraire, en contient toujours, de part et d’autre de la séquence visée. Moineau essaie de les retirer, la défense cesse d’opérer. Leur raison d’être, conclut-il ? Eviter que Crispr, dans une poussée suicidaire, ne s’attaque à l’ADN de la bactérie elle-même. Car c’est bien l’ADN qui est directement visé par le système. Moineau et ses collègues en apportent la preuve, en 2010, par une observation directe, cette fois.
Il manque encore une dernière pièce. Française installée à Vienne, Emmanuelle Charpentier l’apporte lors de la réunion Crispr d’octobre 2010, à Wageningen (Pays-Bas). Elle y annonce qu’un autre type de petit ARN (baptisé tracrRNA) est indispensable aux bactéries pour couper leur ARN et lancer la chasse aux virus décrite par van der Oost deux ans auparavant. Ses résultats seront publiés dans Nature en 2011.
Le puzzle est désormais prêt, il reste à l’assembler. Sortir le système de la bactérie pour réaliser ce que tout le monde commence à entrevoir : un outil d’édition du génome, dont personne ne mesure toutefois la portée. C’est le sprint final. Sauf qu’à ce petit jeu, tous ne partent pas à égalité. D’abord parce que chaque bactérie a son Crispr, plus ou moins sophistiqué. Escherichia coli, chère à John van der Oost, utilise un complexe de cinq protéines. Staphylococcus epidermidis, qu’étudient Marraffini et Sontheimer, pas moins de neuf. A l’inverse, Charpentier, avec Streptococcus pyogenes, et Moineau, avec son S. thermophilus, ont hérité d’un Crispr dit de type 2, avec une seule et unique protéine : Cas9. « Là, c’est sûr, on a eu un peu de chance », admet la chercheuse française.
Enfin il y a les hasards des intérêts, des carrières et des financements. Moineau le dit sans détour : « Je n’ai pas mesuré le potentiel. » « J’étais un peu trop occupé à monter mon propre labo », dit Luciano Marraffini, aujourd’hui à l’université Rockefeller, à New York. A Chicago, Erik Sontheimer veut basculer sur le type 2. Mais en cette période de « vaches maigres », il peine à embaucher des postdocs et à disposer du matériel nécessaire. Dès janvier 2009, il a fait une demande de bourse aux National Institutes of Health sur le créneau « haut risque, haut potentiel ». Il essuie un refus. Ses demandes en mars et novembre 2010 auprès de la Fondation Bill Gates resteront aussi infructueuses : « J’en ai décroché beaucoup d’autres dans ma carrière, mais c’était les trois meilleurs dossiers et ils ont été rejetés… »
En juin 2012, le coup de tonnerre finit par retentir. Science publie le fruit d’une collaboration entre les laboratoires d’Emmanuelle Charpentier et de la biologiste de l’université de Californie à Berkeley Jennifer Doudna. Elles y décrivent la première reconstitution de Crispr-Cas9 in vitro. Une protéine unique, deux ARN programmables – et même un seul, grâce à une astucieuse manipulation afin de choisir sa cible : la possibilité d’intervenir sur le génome de différents types de cellules avec une facilité et une précision jusqu’ici inégalée, assurent-elles. Deux étoiles viennent de naître, prêtes à partir à l’assaut du firmament.